Python OpenCV分水岭分割算法
求则得之,舍则失之 人气:0前言
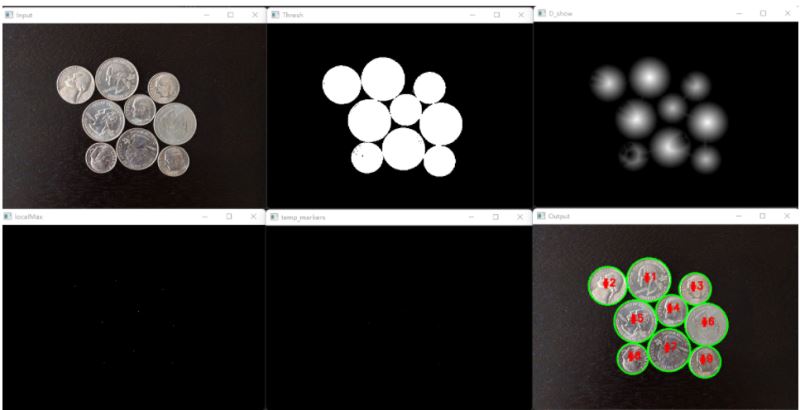
分水岭算法是用于分割的经典算法,在提取图像中粘连或重叠的对象时特别有用,例如下图中的硬币。

使用传统的图像处理方法,如阈值和轮廓检测,我们将无法从图像中提取每一个硬币,但通过利用分水岭算法,我们能够检测和提取每一个硬币。
在使用分水岭算法时,我们必须从用户定义的标记开始。这些标记可以通过点击手动定义,或者我们可以使用阈值和/或形态学操作等方法自动或启发式定义它们。
基于这些标记,分水岭算法将输入图像中的像素视为地形——该方法通过“淹没”山谷,从标记开始向外移动,直到不同标记相遇。为了获得准确的分水岭分割,必须正确放置标记。
在这篇文章的剩下部分,我将向您展示如何使用分水岭算法来分割和提取图像中既粘连又重叠的对象。
为此,我们将使用各种 Python 包,包括 SciPy、scikit-image 和 OpenCV。
在上图中,您可以看到使用简单阈值和轮廓检测无法提取对象,由于这些对象是粘连的、重叠的或两者兼有,
因此简单的轮廓提取会将粘连的对象视为单个对象,而不是多个对象。
1.使用分水岭算法进行分割
# 打开一个新文件,将其命名为 watershed.py ,然后插入以下代码:
# 打开一个新文件,将其命名为 watershed.py ,然后插入以下代码:
# 导入必要的包
from skimage.feature import peak_local_max
from skimage.morphology import watershed
from scipy import ndimage
import numpy as np
import argparse
import imutils
import cv2
# 构造参数解析并解析参数
ap = argparse.ArgumentParser()
# ap.add_argument("-i", "--image", default="HFOUG.jpg", help="path to input image")
ap.add_argument("-i", "--image", default="watershed_coins_01.jpg", help="path to input image")
args = vars(ap.parse_args())
# 加载图像并执行金字塔均值偏移滤波以辅助阈值化步骤
image = cv2.imread(args["image"])
shifted = cv2.pyrMeanShiftFiltering(image, 21, 51)
cv2.imshow("Input", image)
# 将图像转换为灰度,然后应用大津阈值
gray = cv2.cvtColor(shifted, cv2.COLOR_BGR2GRAY)
thresh = cv2.threshold(gray, 0, 255, cv2.THRESH_BINARY | cv2.THRESH_OTSU)[1]
cv2.imshow("Thresh", thresh)
# 计算从每个二进制图像中的像素到最近的零像素的精确欧氏距离,然后找出这个距离图中的峰值
D = ndimage.distance_transform_edt(thresh)
# 可视化距离函数
D_show = cv2.normalize(D, None, 0, 1, cv2.NORM_MINMAX)
# print(np.max(D_show))
cv2.imshow("D_show", D_show)
# 以坐标列表(indices=True)或布尔掩码(indices=False)的形式查找图像中的峰值。峰值是2 * min_distance + 1区域内的局部最大值。
# (即峰值之间至少相隔min_distance)。此处我们将确保峰值之间至少有20像素的距离。
localMax = peak_local_max(D, indices=False, min_distance=20, labels=thresh)
# 可视化localMax
temp = localMax.astype(np.uint8)
cv2.imshow("localMax", temp * 255)
# 使用8-连通性对局部峰值进行连接成分分析,然后应用分水岭算法
# scipy.ndimage.label(input, structure=None, output=None)
# input :待标记的数组对象。输入中的任何非零值都被视为待标记对象,零值被视为背景。
# structure:定义要素连接的结构化元素。对于二维数组。默认是四连通, 此处选择8连通
#
markers = ndimage.label(localMax, structure=np.ones((3, 3)))[0] # [0, 1, 2, 3, 4, 5, 6, 7, 8, 9]
# 可视化markers
temp_markers = markers.astype(np.uint8)
cv2.imshow("temp_markers", temp_markers * 20)
# 由于分水岭算法假设我们的标记代表距离图中的局部最小值(即山谷),因此我们取 D 的负值。
labels = watershed(-D, markers, mask=thresh)
print("[INFO] {} unique segments found".format(len(np.unique(labels)) - 1))
# 循环遍历分水岭算法返回的标签
for label in np.unique(labels):
# 0表示背景,忽略它
if label == 0:
continue
# 否则,为标签区域分配内存并将其绘制在掩码上
mask = np.zeros(gray.shape, dtype="uint8")
mask[labels == label] = 255
# 在mask中检测轮廓并抓取最大的一个
cnts = cv2.findContours(mask.copy(), cv2.RETR_EXTERNAL,
cv2.CHAIN_APPROX_SIMPLE)
cnts = imutils.grab_contours(cnts)
c = max(cnts, key=cv2.contourArea)
# 在物体周围画一个圆
((x, y), r) = cv2.minEnclosingCircle(c)
cv2.circle(image, (int(x), int(y)), int(r), (0, 255, 0), 2)
cv2.putText(image, "#{}".format(label), (int(x) - 10, int(y)),
cv2.FONT_HERSHEY_SIMPLEX, 0.6, (0, 0, 255), 2)
# 显示输出图像
cv2.imshow("Output", image)
cv2.waitKey(0)
2.Watershed与random walker分割对比
示例比较了两种分割方法,以分离两个相连的磁盘:分水岭算法和随机游走算法。 两种分割方法都需要种子,即明确属于某个区域的像素。在这里,到背景的距离图的局部最大值被用作种子。
import numpy as np
from skimage.segmentation import watershed
from skimage.feature import peak_local_max
from skimage import measure
from skimage.segmentation import random_walker
import matplotlib.pyplot as plt
from scipy import ndimage
# import cv2
# Generate an initial image with two overlapping circles
x, y = np.indices((80, 80))
x1, y1, x2, y2 = 28, 28, 44, 52
r1, r2 = 16, 20
mask_circle1 = (x - x1) ** 2 + (y - y1) ** 2 < r1 ** 2
mask_circle2 = (x - x2) ** 2 + (y - y2) ** 2 < r2 ** 2
image = np.logical_or(mask_circle1, mask_circle2)
# Now we want to separate the two objects in image
# Generate the markers as local maxima of the distance
# to the background
distance = ndimage.distance_transform_edt(image)
D_show = distance/np.max(distance)
# D_show = cv2.normalize(distance, None, 0, 1, cv2.NORM_MINMAX)
local_maxi = peak_local_max(
distance, indices=False, footprint=np.ones((3, 3)), labels=image)
markers = measure.label(local_maxi)
labels_ws = watershed(-distance, markers, mask=image)
markers[~image] = -1
labels_rw = random_walker(image, markers)
plt.figure(figsize=(12, 3.5))
plt.subplot(141)
plt.imshow(image, cmap='gray', interpolation='nearest')
plt.axis('off')
plt.title('image')
plt.subplot(142)
plt.imshow(D_show, cmap='Spectral',interpolation='nearest')
plt.axis('off')
plt.title('distance map')
plt.subplot(143)
plt.imshow(labels_ws, cmap='Spectral', interpolation='nearest')
plt.axis('off')
plt.title('watershed segmentation')
plt.subplot(144)
plt.imshow(labels_rw, cmap='Spectral', interpolation='nearest')
plt.axis('off')
plt.title('random walker segmentation')
plt.tight_layout()
plt.show()

加载全部内容