python 服务器批处理
李划水员 人气:01. 在linux上安装psiblast
最好新建一个python环境,因为我发现conda安装blast默认的是python==3.6.11,可能会不小心把你的python版本改掉…然后你写好的代码全die了……
conda create -n blast python==3.6.11 source activate blast conda install -c bioconda blast
2.下载并编译用于比对的大型蛋白质数据库
nr和uniprot是比较通用的数据库:
ftp://ftp.ncbi.nlm.nih.gov/blast/db/
https://www.uniprot.org/downloads
1)nr是ncbi收集的目前所有微生物的蛋白序列,是用来计算氨基酸一般情况下的频率的,160G
2)uniprot90根据相似性做了一个去冗余,所以比nr要小很多,56G
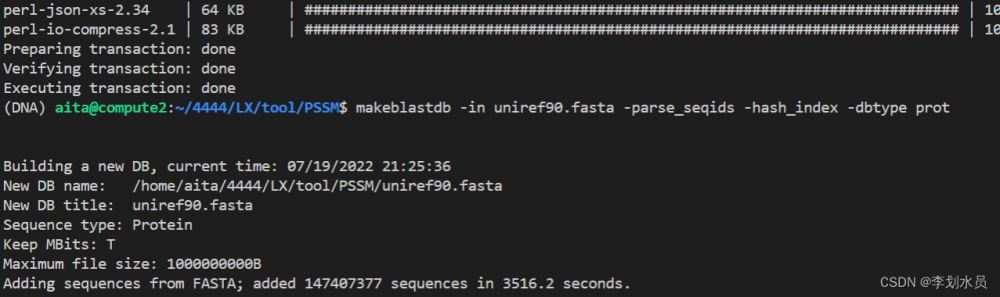
# 以uniprot90为例 wget ftp://ftp.uniprot.org/pub/databases/uniprot/uniref/uniref90/uniref90.fasta.gz # 下载 gzip -d uniref90.fasta.gz # 解压 makeblastdb -in uniref90.fasta -parse_seqids -hash_index -dbtype prot # 编译
解析完成后的样子:
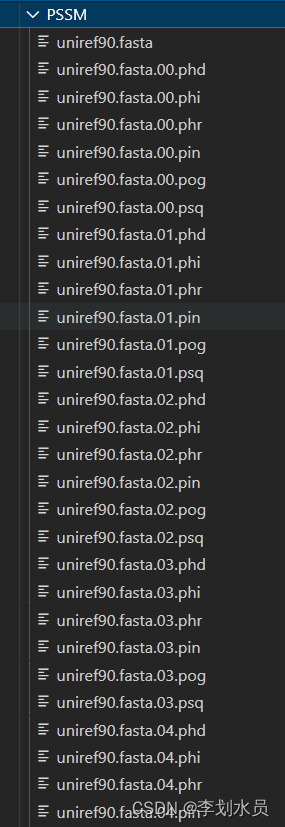
文件是这个样子:(只截取了一部分)
3. 获取PSSM矩阵
我的初始文件是:
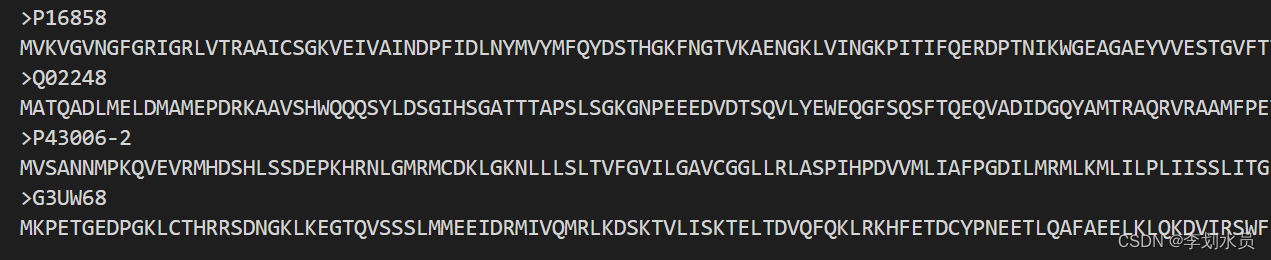
P00269.fasta是对单条蛋白质处理,里面的格式是:

testset.fasta是对蛋白质集合批处理,里面的格式是(也可以单独蛋白质存为.fasta文件,由于blast只能处理单条蛋白糊,把这个集合知识归总的意思,第一步还是要生成单条蛋白质的.fasta文件,所以这个文件看个人意愿):
1)单条蛋白质序列的处理方法
import os
os.system('psiblast -query dataset/P00269.fasta -db /PSSM/uniref90.fasta -num_iterations 3 -out_ascii_pssm /dataset/P00269.pssm')##这个蛋白质好慢呀2)批处理获取的方法
import os
file_name='/dataset/testset.fasta'
Protein_id=[]
with open(file_name,'r') as fp:
i=0
for line in fp:
if i%2==0:
# Protein_id.append(line[1:-1])
id=line[0:-1]
p=line[1:-1]
with open ('/dataset/'+str(p)+'.fasta','a') as protein:
protein.write(id)
# protein.write()
if i%2==1:
seq=line[0:-1]
with open ('/dataset/'+str(p)+'.fasta','a') as protein:
protein.write('\n')
protein.write(seq)
i=i+1
os.system('psiblast -query '+'/dataset/'+str(p)+'.fasta -db /PSSM/uniref90.fasta -num_iterations 3 -out_ascii_pssm /dataset/'+str(p)+'.pssm')##PSSM真是太慢了,下面是只生成一个后的截图
emmmm,在研究怎么把这个矩阵存入文件方便调用,今天应该会更新……但是他好慢啊,不想用了。
参考文献:
加载全部内容